Designprinzipien und Ökonomie zellulärer Informationsverarbeitung
Forschungsbericht (importiert) 2010 - Max-Planck-Institut für Kolloid- und Grenzflächenforschung
Einführung
Die meisten Prozesse, die in Zellen ablaufen, werden sehr genau kontrolliert. Zum Beispiel gibt es zahlreiche Regulationsmechanismen, die Gene an- oder ausschalten oder deren Aktivität genau einstellen können. Da die Proteine dieser Steuerungsmechanismen ebenfalls von ihren jeweiligen Genen codiert werden, werden Gene effektiv von anderen Genen reguliert. Die komplexen Netzwerke gegenseitiger Regulation werden oft mit elektronischen Schaltkreisen verglichen. Welche Prinzipien dem Design solcher genetischen Schaltkreise zugrunde liegen, welchen physikalischen Zwangsbedingungen sie unterworfen sind, und wie synthetische genetische Schaltkreise konstruiert werden können, sind Fragen, die aktuell untersucht werden. Theoretische Untersuchungen spielen in diesem Zusammenhang eine wichtige Rolle, da sie eine Brücke von der molekularen Beschreibung eines Systems zu dessen physiologischem oder evolutionären Kontext schlagen können.
Molekulare Maschinen der genetischen Information und ihre Ökonomie
Um die genetische Information auszulesen, besitzen Zellen ein ganzes Arsenal molekularer Maschinen. Im einfachsten Fall wird die DNA-Sequenz eines Gens in einem ersten Schritt von einer RNA-Polymerase (RNAP) in ein RNA-Molekül transkribiert. Dieses wird dann von einem Ribosom ausgelesen, welches die der Gensequenz entsprechende Proteinkette synthetisiert. Die Verfügbarkeit dieser Maschinen spielt eine wichtige Rolle für die genetische Informationsverarbeitung. Ihre Konzentrationen sind umso höher, je schneller die Zellen wachsen; besonders stark ausgeprägt ist dies in exponentiell wachsenden Bakterienkulturen [1].
Die Abhängigkeit der zellulären Konzentrationen von Ribosomen und RNAPs von den Wachstumsbedingungen bietet einen Ansatzpunkt, um die ökonomischen Prinzipien zu untersuchen, die dem Einsatz dieser Maschinen in der Zelle zugrunde liegen. Überraschenderweise sind diese Prinzipien für die beiden Arten von molekularen Maschinen der Genexpression sehr unterschiedlich. Ribosomen werden von Bakterienzellen wie sehr teure Investitionen behandelt, die möglichst effizient genutzt werden müssen. Daher sind, zumindest bei schnellem Wachstum, zu jedem Zeitpunkt nur sehr wenige Ribosomen frei verfügbar, während die meisten mit der Synthese von Proteinen beschäftigt sind. Diese Beobachtung erlaubt es, evolutionäre Überlegungen anzustellen, in denen eine höhere Auslastung der Ribosomen einen Fitness-Vorteil darstellt.
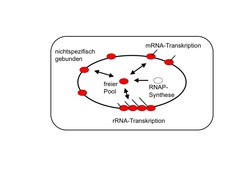
Die Anzahl der RNAPs in einer Bakterienzelle ist dagegen deutlich größer als die Anzahl an RNAPs, die gleichzeitig zur Transkription benötigt werden. Ein großer Teil der RNAPs ist unspezifisch an die DNA gebunden. Wie Zellen RNAPs zwischen verschiedenen Klassen von Genen, nichtspezifischer DNA-Bindung und dem Pool freier RNAPs aufteilen, kann mit einem physikalischen Modell für Bindung und Transkription untersucht werden (Abb. 1) [2]. Da die Parameter von Bakterienzellen unter verschiedenen Wachstumsbedingungen seit den späten 50er-Jahren sehr ausführlich untersucht wurden [1], konnten alle Parameter dieses Modells bestimmt werden, so dass quantitative Vorhersagen möglich sind. Unter anderem konnte mit diesem Modell bestimmt werden, wie der Pool der freien RNAPs, welcher die Verfügbarkeit für die Transkription festlegt, von der bakteriellen Wachstumsrate abhängt.
Transkription ribosomaler RNA
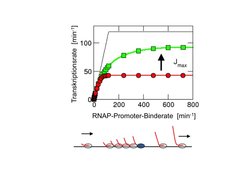
Die Transkription der ribosomalen RNA (rRNA), der RNA-Bestandteile der Ribosomen, hat eine herausragende Bedeutung in der Zelle; in schnell wachsenden Zellen dominiert sie nämlich die gesamte Transkription [3]. Plötzliche Änderungen in der Transkription der rRNA könnten daher zu großen Änderungen in der Anzahl der frei verfügbaren RNAPs führen, was die Transkription aller Gene beeinflussen würde. Ob dies so geschieht, ist ebenfalls mit dem physikalischen Modell für die RNAP-Aufteilung untersucht worden. Diese Berechnungen zeigen, dass die nichtspezifisch gebundenen RNAPs hier als Puffer wirken, der die Gene gegen solche dramatischen Veränderungen absichert [2]. Die Transkription der rRNA unterscheidet sich von der sonstigen Transkription auch durch einen besonders dichten Verkehr der RNAPs; sie unterliegt daher besonderen physikalischen Zwangsbedingungen, die mit Transportmodellen aus der Statistischen Physik des Nichtgleichgewichts theoretisch untersucht werden können [4, 5]. Zum Beispiel müssen Pausen in der Transkription stark unterdrückt sein, um Staus zu verhindern, da Staus die Transkriptionsrate der rRNA reduzieren und das Zellwachstum beeinträchtigen können (Abb. 2) [4].
Genetische Schaltkreise unter variablen Zellwachstumsbedingungen
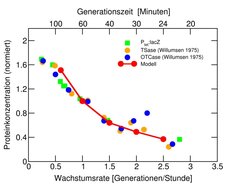
Da die Verfügbarkeit der molekularen Maschinen für die Transkription und Translation vom physiologischen Zustand der ganzen Zelle abhängt, sind die Aktivitäten aller Gene und die Funktionen aller natürlichen und synthetischen genetischen Schaltkreise an den physiologischen Zustand der Zelle gekoppelt. Diese Kopplung stellt eine Komplikation für die Konstruktion synthetischer genetischer Schaltkreise dar, die für bakterielle Systeme quantitativ untersucht wurde. Für diesen Fall sind alle elementaren Parameter der Genexpression in Abhängigkeit von der Wachstumsrate der Bakterien gemessen worden, sodass die Wachstumsabhängigkeit der Produkte eines Gens mithilfe eines Modells theoretisch vorhergesagt werden kann [6]. Diese Vorhersagen wurden für einige einfache Schaltkreise experimentell bestätigt (Abb. 3) [6].
Wenn die Konzentration eines Proteins das Zellwachstum beeinflusst und gleichzeitig das Zellwachstum die Konzentration dieses Proteins, kommt es zu einer Rückkopplung, die durch Änderungen des Zellwachstums vermittelt wird. Diese kann entweder unabhängig von speziellen Regulationsmechanismen wirken [6] oder eine auf Regulation basierte Rückkopplung verstärken [7]. Im Fall einer positiven Rückkopplung, wenn z.B. ein Protein das Zellwachstum verlangsamt, langsamer wachsende Zellen aber höhere Konzentrationen diese Proteins enthalten, kann es zur Bistabilität kommen: In diesem Fall spaltet sich eine Zellpopulation in zwei Subpopulationen, die unterschiedlich schnell wachsen. Es wurde vorgeschlagen, dass ein solcher Rückkopplungsmechanismus bei der Ausbildung von Antibiotikatoleranz von Bakterien (Persistenz) eine Rolle spielt [6].
Zusammenfassung und Ausblick
In den letzten Jahren hat das quantitative Verständnis von genetischen Schaltkreisen große Fortschritte gemacht. Die Kopplung dieser Schaltkreise an den physiologischen Gesamtzustand der Zelle erschwert zwar die Konstruktion von synthetischen Schaltkreisen und deren Standardisierung, sie ermöglicht aber auch einen Einblick in die Gesetzmäßigkeiten, die natürlichen genetischen Schaltkreisen zugrunde liegen, und in die ökonomischen Strategien, mit denen Zellen molekulare Maschinen einsetzen.


