Semiflexible Polymere und Filamente: Vom einzelnen Polymer zur aktiven biologischen Struktur
Forschungsbericht (importiert) 2006 - Max-Planck-Institut für Kolloid- und Grenzflächenforschung
Einleitung
Viele Biopolymere, wie die DNS oder die fadenartigen Polymere des Zellskeletts, sind semiflexibel, d.h., sie besitzen eine mechanische Biegesteifigkeit, die Form und Fluktuationen dieser Polymere bestimmt. Die Biegesteifigkeit ist in der Regel durch die biologische Funktion dieser Polymere bedingt. So bilden die fadenartigen Filamente das Skelett eukaryotischer Zellen, das einerseits dazu dient, die Zellform
aufrecht zu erhalten und der Zelle mechanische Stabilität zu verleihen, andererseits aber Bewegung und Formveränderung von Zellen ermöglichen muss. Auch für die Organisation der DNS im Erbgut ist eine gewisse Biegesteifigkeit wesentlich, die zufällige Konformationsänderungen unterdrückt. Daneben spielen synthetische semiflexible Polymere auch in der chemischen Physik eine wichtige Rolle. So besitzen Polyelektrolyte, also geladene Polymere, aufgrund der abstoßenden elektrostatischen Wechselwirkung zwischen gleichen Ladungen eine vergrößerte Kettensteifigkeit, die durch die Ladungsdichte auf dem Polymer und den Salzgehalt der umgebenden Lösung bestimmt ist. Polyelektrolyte stellen eine wichtige Substanzklasse in der Polymerchemie dar.
Die Filamente des Zytoskeletts geben biologischen Zellen nicht nur mechanische Stabilität, sie spielen auch eine große Rolle für intrazellulären Transport und Dynamik. Auf den Filamenten kann die Bewegung von Motorproteinen gerichtet verlaufen, was einen schnellen Transport über große intrazelluläre Entfernungen entlang der „Filamentschienen“ des Zytoskeletts ermöglicht. Neben ihrer Funktion als Nano-Zugmaschinen sind molekulare Motoren auch an der ständigen Umorganisation des Zytoskeletts selbst aktiv beteiligt. Darüber hinaus baut sich das Zytoskelett auch durch Polymerisation und Depolymerisation von Filamenten permanent um. Sowohl die Bewegung von Motorproteinen als auch die Filamentpolymerisation werden chemisch angetrieben durch Hydrolyse von ATP und sind daher Ursache dynamischer Umordnungsprozesse fern vom Gleichgewicht.
Semiflexible Polymere stellen damit sowohl vom Standpunkt der Polymer- als auch als vom Standpunkt der biologischen Physik interessante Systeme dar. Ziel der vorgestellten Arbeiten ist es, die Materialeigenschaften semiflexibler Polymere zu charakterisieren und das Verhalten von Filamenten in komplexen biologischen Systemen zu verstehen. Dazu werden Systeme untersucht, die von einzelnen Polymeren bis zu wechselwirkenden Filamenten im Zusammenspiel mit Motorproteinen reichen. Dabei ist es notwendig, Einfluss und Wechselspiel thermischer Fluktuationen, externer Kräfte, der Wechselwirkungen zwischen Filamenten und chemisch getriebener aktiver Prozesse zu verstehen.
Die Physik einzelner semiflexibler Polymere
Ein anschauliches Maß für die Biegesteifigkeit ist die Persistenzlänge eines Polymers, die eine Längenskala angibt, auf der sich ein semiflexibles Polymer aufgrund thermischer Kräfte verformen kann. Die Persistenzlänge wird durch das Verhältnis von Biegesteifigkeit κ und thermischer Energie bestimmt. Die Biegeenergie koppelt thermische Fluktuationen auf verschiedenen Längenskalen. Daraus resultiert eine effektiv kleinere Biegesteifigkeit auf großen Längenskalen. Dieser Effekt kann dazu verwendet werden, eine systematische Definition der Persistenzlänge als charakteristische Skala für den Zerfall der Biegesteifigkeit zu entwickeln [1]. Das Verhalten semiflexibler Polymere unterscheidet sich qualitativ von dem flexibler Polymere auf Längenskalen unterhalb dieser Persistenzlänge. Typische Persistenzlängen von Biopolymeren bewegen sich im Bereich von 50 nm für DNS; Filamente des Zytoskeletts haben große Persistenzlängen, die für Aktinfilamente im μm-Bereich liegen und für Mikrotubuli sogar im mm-Bereich.
Das charakteristische Verhalten semiflexibler Polymere kann in Manipulationsexperimenten an einzelnen Polymeren beobachtet werden, die im Laufe der letzten zehn Jahre durch neue Mikromanipulationstechniken basierend auf optischen Pinzetten oder dem Rasterkraft-Mikroskop möglich wurden. Um solche Einzelpolymer-Experimente quantitativ auszuwerten, bedarf es theoretischer Modelle. Beispiele dafür sind die Fluktuationsanalyse von einzelnen Aktinfilamenten in Mikrokanälen [2], die Manipulation von Polymeren mithilfe von Mikroskop-Spitzen [3, 4], oder die kraftinduzierte Desorption einzelner Polymere von einer adhäsiven Oberfläche, wie sie schematisch in Abbildung 1 dargestellt ist. Bei dem letztgenannten System zeigt sich beispielsweise, dass die Biegesteifigkeit eines adsorbierten Polymers zu einer Energiebarriere führt, die überwunden werden muss, bevor es von der adsorbierenden Oberfläche abgezogen werden kann [5].

Filamentbündel
Biologische Zellen sind mechanisch stabil, da sie Aktinfilamente und Mikrotubuli besitzen, die Netzwerke und Bündel ausbilden und ein stützendes Zellskelett aufbauen. Diese Architekturen werden von quervernetzenden Proteinen, die zwei adhäsive Enden besitzen und damit verschiedene Filamente aneinander binden können, zusammengehalten und kontrolliert. Will man die für diese Prozesse verantwortlichen Kräfte verstehen, um beispielsweise die mechanischen Eigenschaften der Architekturen zu optimieren, muss man biomimetische Modellsysteme studieren, die allein aus Filamenten und quervernetzenden Proteinen bestehen. Ein wichtiges Beispiel dafür ist die Vernetzung mehrerer Filamente zu dicken Bündeln, die steifer sind und eine größere externe Last tragen können als ein einzelnes Filament.
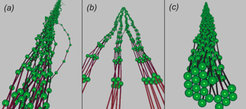
Die Vernetzung von Filamenten wird durch ihre thermische Bewegung verhindert, sofern die Konzentration der Vernetzer einen bestimmten kritischen Schwellwert nicht überschreitet [6]. Dieser Schwellwert hängt von der Filamentsteifigkeit, der Bindungsenergie der quervernetzenden Moleküle und der Temperatur ab. Die kritische Vernetzerkonzentration nimmt ab, wenn die Anzahl N der Filamente innerhalb des Bündels zunimmt, bleibt aber endlich im Limes großer N. Oberhalb der kritischen Vernetzerkonzentration findet ein diskontinuierlicher Phasenübergang statt, bei dem sich ein Bündel zusammenschließt. Dieser Mechanismus kann auch in der Zelle genutzt werden, um Bündelbildung durch die Vernetzerkonzentration im Zytoplasma zu kontrollieren. Die Abbildung 2 zeigt Schnappschüsse von Filamentbündeln, wie sie in der Computersimulation beobachtet werden. Dabei wird klar, dass sich große Bündel oft auftrennen in kleinere Subbündel und die Bündelmorphologie von der Gleichgewichtsform abweicht. Dies liegt daran, dass für große Bündel die Kinetik des Vernetzungsprozesses und die anfängliche Anordnung der Filamente eine große Rolle spielen.
Abgesehen von ihrer Aufgabe als strukturelle Elemente können Filamentbündel auch Druckkräfte erzeugen. Diese entstehen durch das gerichtete Wachstum der Filamente, wenn weitere molekulare Bausteine an den Filament-Enden angebaut werden. In der Zelle wird dieses Wachstum durch ATP- oder GTP-Hydrolyse verstärkt und kontrolliert und wird zur Krafterzeugung genutzt, z.B. bei der Zellbewegung. Computersimulationen zeigen, dass es für Bündel von Filamenten neben diesem Polymerisationsmechanismus noch einen alternativen Mechanismus der Krafterzeugung gibt [7]. Dabei wird die Adhäsionsenergie, die bei der Bündelbildung freigesetzt wird, dazu genutzt, eine Druckkraft zu erzeugen.
Aktive Filamente
Auf den Filamenten des Zytoskeletts kann intrazellulärer Transport durch gerichtete Bewegung von Motorproteinen schneller und effektiver ablaufen als z.B. durch rein diffusive Prozesse. Daneben sind molekulare Motoren aber auch an der ständigen Umorganisation des Zytoskeletts selbst aktiv beteiligt. Diese Umorganisation ist notwendig für Zellbewegung und Zellteilung. Während dieser Prozesse sind die Filamente des Zytoskeletts in ständiger Bewegung, trotzdem werden in diesem dynamischen Zustand geordnete Strukturen wie die mitotische Spindel hervorgebracht. Um die Prinzipien zu verstehen, die hinter dieser motorgetriebenen Dynamik und den damit verbundenen Musterbildungsprozessen durch Filamente des Zytoskeletts stehen, können Modellsysteme wie „Motility-Assays“ betrachtet werden, die nur einige wenige Komponenten enthalten.
In einem Motility-Assay werden molekulare Motoren auf einer Oberfläche adsorbiert und verankert, über die sie Zytoskelett-Filamente ziehen. Unter dem Einfluss dieser Zugbewegung und im Zusammenspiel mit Kollisionen beginnen die Filamente, sich spontan parallel zu ordnen, wie die Simulationsschnappschüsse in Abbildung 3 zeigen [8, 9]. Diese parallele Ordnung von Filamenten ist der so genannten nematischen Ordnung stäbchenförmiger Moleküle in einem Flüssigkristall sehr ähnlich. Aber während die parallele Ordnung im Flüssigkristall nur durch eine Erhöhung der Filamentdichte oder -länge ausgelöst werden kann, kann die Filamentordnung im Motility-Assay auch durch eine Erhöhung der Motorendichte erreicht werden. Überraschenderweise verstärkt somit die motorgetriebene Bewegung der Filamente die Tendenz zur Ordnung. Dies scheint einem physikalischen Grundprinzip zu widersprechen, demzufolge mikroskopische Bewegung von Molekülen im Allgemeinen zu einer Zerstörung ihrer Ordnung führt, wie z.B. beim Schmelzen eines Kristalls.
Die größere Orientierungsordnung der Filamente unter dem Einfluss der Aktivität molekularer Motoren kann in Computersimulationen gezeigt werden, wie sie in Abbildung 3 zu sehen sind. In Abbildung 3(a) ist die Motordichte klein und die Filamente zeigen keine Ordnung. In Abbildung 3(b) wurde lediglich die Motorendichte erhöht und die Filamente ordnen sich parallel zueinander an. Theoretisch kann dieser Effekt mithilfe des Konzeptes einer effektiven Filamentlänge erklärt werden, die sich vergrößert aufgrund der Motoraktivität: Weil die Motoren Filamente entlang ihrer Achse bewegen, haben diese eine größere effektive Länge und beginnen bereits bei geringeren Dichten, miteinander wechselzuwirken und sich dadurch parallel anzuordnen [8]. Dieses allgemeingültige theoretische Konzept sollte sich auch auf andere Motor-Filament-Systeme anwenden lassen.



