Den verschiedenen Tempi der Proteinsynthese auf der Spur
Forscher der Max-Planck-Institute für Kolloid- und Grenzflächenforschung in Potsdam und für biophysikalische Chemie in Göttingen haben eine Methode entwickelt, mit der sich die unterschiedlichen, codon-spezifischen Geschwindigkeiten der Proteinsynthese in lebenden Zellen aus in-vitro-Experimenten bestimmen lassen.
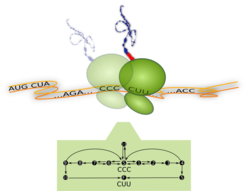
Ribosom bei der Proteinsynthese und Prozessdiagramm:
(oben) Das Ribosom (grün) ist eine biomolekulare Maschine mit einem Durchmesser von etwa 23 nm. Es bewegt sich entlang der Boten-RNA (Band in orange) und übersetzt dabei ein Codon (..., CCC, CUU, ...) nach dem anderen in jeweils eine Aminosäure. Die neue Aminosäure (rot) wird an die wachsende Peptidkette (blau) angehängt.
(unten) Prozessdiagramm mit internen Reaktionsschritten (schwarze Pfeile) des Ribosoms bei der Übersetzung des Codons CCC. Das Ribosom startet im internen Zustand S (Start) und kann drei Reaktionspfade durchlaufen. Diese entsprechen der Bindung von verschiedenen Transfer-RNA-Molekülen (nicht dargestellt). Die Übersetzung des Codons ist abgeschlossen, sobald das Ribosom den molekularen Zustand F (Finish) erreicht und sich zum nächsten Codon, hier CUU, bewegt hat. Ist das Ribosom in einem internen Zustand, von dem mehrere Pfade abgehen, kann es diesen mit unterschiedlichen Wahrscheinlichkeiten folgen.
Leben bedeutet Bewegung – das gilt sowohl für die makroskopische als auch für die mikroskopische Welt. Könnten wir tief ins Innere einer lebenden Zelle schauen, so würden wir beobachten, dass ständig neue zelluläre Komponenten produziert, verändert, transportiert und schließlich wieder abgebaut werden. Diese dynamischen Prozesse werden von einer Vielzahl winziger biomolekularer Maschinen angetrieben. Ein eindrucksvolles Beispiel dafür ist die Proteinsynthese durch Ribosomen (Abb. 1). Mit einem Durchmesser von etwa 23 Nanometern ist das Ribosom ein echter „Nanoroboter“. Während der Proteinsynthese bewegt es sich entlang einer Boten-RNA, die aus einer Kette von Codons besteht. Als Codon bezeichnet man die Sequenz von drei Nukleobasen der mRNA, die im genetischen Code eine Aminosäure codiert. Das Ribosom übersetzt diese Codonsequenz der Boten-RNA in die Aminosäuresequenz des zugehörigen Proteins. Sobald das Ribosom an einem neuen Codon angekommen ist, sucht es nach einer geeigneten Transfer-RNA, die mit der richtigen Aminosäure beladen ist. Dabei durchläuft es eine Folge von internen Transformationen, (Prozessdiagram in Abb. 1) und verlängert die wachsende Peptidkette des Proteins um eine Aminosäure. Anschließend bewegt sich das Ribosom zum nächsten Codon, um auch dieses in eine Aminosäure zu übersetzen. Dieser Prozess setzt sich fort, bis das neue Protein fertiggestellt ist.
Eine zuverlässige und präzise Proteinsynthese durch die Ribosomen ist entscheidend für alle Lebensvorgänge. Denn nicht nur unsere Zellen, auch die aller anderen Organismen bestehen zum größten Teil aus Proteinen. Wird die Synthese neuer Proteine verhindert, kann eine Zelle nicht mehr richtig funktionieren und stirbt. So setzt die Behandlung von Infektionskrankheiten auch genau hier an: Mit Antibiotika werden die einzelnen Schritte der Proteinsynthese in den Bakterien gezielt blockiert und die Mikroorganismen dadurch getötet.
Ribosomen arbeiten allerdings nicht wie herkömmliche Maschinen. Sie können verschiedenen, zufällig ausgewählten Reaktionspfaden folgen (Prozessdiagramm in Abb. 1). Die verschiedenen Reaktionspfade entsprechen der Anbindung verschiedener Transfer-RNA-Moleküle, die in unterschiedlicher Menge in den Zellen vorhanden sind. Das Ribosom probiert deshalb viele Transfer-RNA-Moleküle mit verschiedenen Aminosäuren aus, bevor es ein geeignetes Molekül „erkennt“. Erst dann „entscheidet“ es sich für die weitere Verarbeitung dieses Moleküls. Außerdem ist die zeitliche Dauer der einzelnen Reaktionsschritte nicht genau festgelegt, sondern bei jedem Durchgang unterschiedlich lang. Bei den einzelnen Reaktionsschritten spielt der Zufall eine große Rolle. Dennoch arbeitet das Ribosom sehr zuverlässig und baut nur selten eine falsche Aminosäure ein. Die unterschiedlichen Reaktionspfade und zufälligen Schrittzeiten haben allerdings eine wichtige Konsequenz: Die verschiedenen Codons werden mit unterschiedlicher Geschwindigkeit übersetzt. Die Forscher der Max-Planck-Institute für Kolloid- und Grenzflächenforschung in Potsdam und für biophysikalische Chemie in Göttingen wollten nun wissen, wie die Übersetzungsgeschwindigkeit von den individuellen Schrittzeiten abhängt und wie stark sich diese Geschwindigkeit für die 61 verschiedenen Codons in lebenden Zellen unterscheidet.
Derzeit ist es noch nicht möglich die Schrittzeiten direkt in lebenden Zellen zu messen, da deren hochkomplexes Inneres nicht mit molekularer Auflösung beobachtet werden kann. Deswegen werden Experimente in vitro durchgeführt: Die Ribosomen werden aus den Zellen herausgenommen und isoliert „im Reagenzglas“ untersucht. „Das in-vitro-Verhalten entspricht oft nicht demjenigen in der Zelle. Man kann sich das so vorstellen wie bei einer Pflanze, die in der Natur ganz anders wächst und gedeiht als daheim im Topf auf der Fensterbank. Es stellt sich daher die Frage, welche Erkenntnisse aus solchen sehr aufwändigen Experimenten gewonnen werden können“, sagt Sophia Rudorf, beteiligte Wissenschaftlerin am MPI für Kolloid- und Grenzflächenforschung.
In der jetzt veröffentlichten Arbeit verknüpfen die Forscher, die über Jahre gewonnenen experimentellen in-vitro-Daten zur Proteinbiosynthese (MPI in Göttingen) mit einem neuen mathematischen Algorithmus (MPI in Potsdam). Dieser überträgt die in-vitro-Ergebnisse auf die Situation in lebenden Zellen. Dazu bestimmt er all diejenigen theoretischen Schrittzeiten, die mit den vorhandenen Erkenntnissen über den Prozess in der Zelle kompatibel sind. Danach bestimmt der Algorithmus von allen theoretisch möglichen Schrittzeiten diejenigen, die „am dichtesten“ an den in vitro gemessenen Schrittzeiten liegen. Dafür musste ein neues Abstandsmaß eingeführt werden, der sogenannte kinetische Abstand. Die so vorhergesagten Schrittzeiten der Proteinbiosynthese wurden erfolgreich durch Vergleich mit bereits vorhandenen Daten zur Geschwindigkeit und Fehleranfälligkeit der Proteinsynthese in Bakterien geprüft. Dabei stellte sich heraus, dass die 61 codon-spezifischen Geschwindigkeiten stark variieren. Die langsamsten und die schnellsten Übersetzungsgeschwindigkeiten der verschiedenen Codons unterscheiden sich um mehr als das Zehnfache.
Die neue Methode erlaubt es, die Dynamik der Proteinsynthese nun auch in lebenden Zellen zu berechnen. „Dies ist nicht nur für die Erforschung von Antibiotika wichtig. Es gibt genetisch verursachte Krankheiten, bei denen die Proteinsynthese erheblich eingeschränkt ist. Die neuen Erkenntnisse könnten uns helfen, diese Krankheiten und ihre Mechanismen zu verstehen“, erläutert Rudorf. Die Methode kann aber auch auf viele andere Zellprozesse angewendet werden, wie z.B. auf die katalytische Aktivität von Enzymen oder auf die Faltung von Proteinen. Viele Krankheiten sind mit diesen Vorgängen ursächlich verbunden. Die zugrunde liegenden Prozesse können aber nur in ihrer natürlichen Umgebung, dem Zellinneren, umfassend verstanden werden.












